Случай повторной инфекции SARS-CoV-2 с геномными данными

Оригинал: The Lancet
Автор: Tillettet et al.
Опубликовано: 12 октября 2020, The Lancet Infectious Diseases
Перевод: Полина Дроздова, Фонд “Не напрасно!”
Редакция: Наталья Белых, Фонд “Не напрасно!”
Резюме
Введение. В настоящий момент неизвестно, насколько хорошо защищает иммунитет, сформированный в результате инфекции SARS-CoV-2. Возможна ли повторная инфекция SARS-CoV-2 не слишком понятно. Мы описываем исследование двух случаев инфекции SARS-CoV-2 у одного пациента.
Методы. 25-летний мужчина, проживавший в округе Уошо (штат Невада, США), дважды попал в поле зрения представителей местного здравоохранения с симптомами вирусной инфекции. Первый раз при тестировании населения в апреле 2020 г., второй — в конце мая - начале июня 2020 г. при обращении за медицинской помощью с последующей госпитализацией. Мазки из носоглотки пациента брали оба раза, и затем ещё дважды. Для подтверждения инфекции SARS-CoV-2 была проведена амплификация нуклеиновых кислот. Мы провели секвенирование нового поколения для вируса SARS-CoV-2, выделенного из мазков из носоглотки. Данные секвенирования проанализированы с помощью двух различных биоинформатических подходов. Маркеры коротких концевых повторов (STR) использованы для фрагментного анализа, чтобы подтвердить, что оба образца инфекций взяты от одного и того же пациента.
Результаты. У пациента было два положительных теста на наличие SARS-CoV-2: 18 апреля 2020 г. и 5 июня 2020 г., разделенных двумя отрицательными, полученными в мае 2020 г.. Анализ генома SARS-CoV-2 показал существенные генетические различия между вариантами, ассоциированными с каждым из случаев инфицирования. Вторая инфекция по симптоматике протекала тяжелее, чем первая.
Интерпретация. Генетические различия двух образцов SARS-CoV-2 были выше, чем можно было бы ожидать при кратковременной эволюции in vivo. Эти результаты позволяют предположить, что пациент был инфицирован SARS-CoV-2 дважды, независимо, генетически различными вирусами. Таким образом, столкновение с SARS-CoV-2 в анамнезе не может гарантировать полный иммунитет от этого вируса. Профилактика заражения SARS-CoV-2 важна для всех одинаково, вне зависимости от того, переболел человек COVID-19 или нет. Возможность повторного инфицирования имеет значение для разработки и применения вакцин.
Введение
Инфекция SARS-CoV-2 приводит к заметному иммунному ответу, но подверженность людей реинфекции SARS-CoV-2 пока не изучена. У зараженных SARS-CoV-2 пациентов вырабатываются нейтрализующие антитела [1]. Тем не менее, пока не выяснено, в какой степени иммунный ответ указывает на развитие иммунитета, защищающего от последующей инфекции SARS-CoV-2. В исследованиях иммунитета к другим коронавирусам [2, 3, 4, 5, 6, 7, 8, 9] защитный иммунитет исчезал в течение 1–3 лет. В случае первичного заболевания со вторичной инфекцией или повторным заболеванием, вызванным тем же возбудителем, разделить события инфицирования с наибольшей точностью позволяет генетический анализ возбудителей, ассоциированных с каждым случаем заболевания. Отчёты о повторных заражениях SARS-CoV-2 были опубликованы исследователями из Гонконга [10], Нидерландов и Бельгии [11] и Эквадора [12]. Мы публикуем клинический случай пациента, который дважды переболел COVID-19, вызванным генетически различными вирусами SARS-CoV-2.
Результаты
Первый мазок из носоглотки, полученный при скрининговом тестировании населения 18 апреля 2020 года показал положительный результат на SARS-CoV-2 при тестировании методом ПЦР с обратной транскрипцией в реальном времени. Два последующих теста методом амплификации нуклеиновых кислот, проведённые после исчезновения симптомов, показали отрицательный результат. Симптомы SARS-CoV-2 вновь проявились у пациента 28 мая 2020 г., и 5 июня 2020 г. он был госпитализирован; в этот момент был получен второй мазок из носоглотки, показавший положительный результат при тестировании на SARS-CoV-2 методом ОТ-ПЦР-РВ. Пациенту была необходима постоянная кислородная поддержка, отмечались миалгия, кашель и одышка. Радиография грудной клетки показала развитие двустороннего интерстициального инфильтрата, позволяющего предположить наличие вирусной или атипичной пневмонии. 6 июня 2020 г. у пациента обнаружены IgG и IgM к SARS-CoV-2 (рис. 1).
Рис. 1. Хронология появления симптомов, молекулярной диагностики и секвенирования. ТОА=транскрипционно-опосредованная амплификация. *Образцы, использованные для секвенирования.
В связи с двумя эпизодами симптоматики, соответствующей COVID-19, и двумя положительными результатами лабораторных исследований на SARS-CoV-2, разделёнными периодом в 48 дней, в течение которых отсутствовали симптомы и было два отрицательных результата теста на SARS-CoV-2, проведено секвенирование нуклеиновой кислоты вирусов, ассоциированных с двумя положительными тестами. Секвенирование по технологии Illumina позволило получить 738 617 пар чтений для образца от апреля 2020 г. (образец A) и 1 410 885 пар чтений для образца от июня 2020 г. (образец B). Данные секвенирования показали, что образец A входит в кладу 20C, поскольку анализ геномной последовательности показал наличие пяти мутаций (однонуклеотидных вариантов (ОНВ)), типичных для клады 20C (3037C→T, 14408C→T, 23403A→G, 1059C→T, and 25563G→T). Образец B также являлся представителем клады 20C и содержал те же пять характерных замен. Образец A содержал ещё пять замен по сравнению с референсным геномом. В образце B были найдены шесть дополнительных ОДВ и мутация в позиции 14 407, соседствующая с 14408C→T и представляющая собой динуклеотидный мультинуклеотидный вариант (МНВ) в позициях 14 407 и 14 408. Шесть ОДВ у образцов A и B совпадали. В образце A были найдены четыре дополнительных ОНВ, не зарегистированных в образце B, в то время как в образце B были найдены семь ОНВ, не найденных в образце А. Соотношение данных секвенирования образцов A и B показано на рис. 2. В образце B были найдены ещё три выпадения нуклеотидов и одна вставка по сравнению с референсным геномом. Эти результаты получили подтверждение при дополнительном анализе файлов FASTQ для соответствующих образцов (не был подтверждён только ОНВ в локусе 4113 в образце A). Предсказания вставок и выпадений были менее стабильны: только выпадение в локусе 2084 и вставка в локусе 6018 были подтверждены. Анализ с помощью алгоритма Freebayes показал выпадение в положении 22 832 в образце B, не идентифицированное при первом анализе последовательностей, но предсказание вставок и выпадений на основе выравнивания коротких чтений менее надёжно, чем предсказание ОНВ [13], и представлено здесь исключительно для полноты описания данных.
Рис. 2. Картирование отличий образцов A и B от референсного генома. ORF1a и ORF1b кодируют белки репликазы. Остальные открытые рамки считывания (ORF, open reading frame) кодируют белки сборки. S=спайк. E=оболочка. M=мембрана. N=нуклеокапсид. *отмечает вариант 14 407 в образце A и варианты 14 407 и 14 408 в образце B.
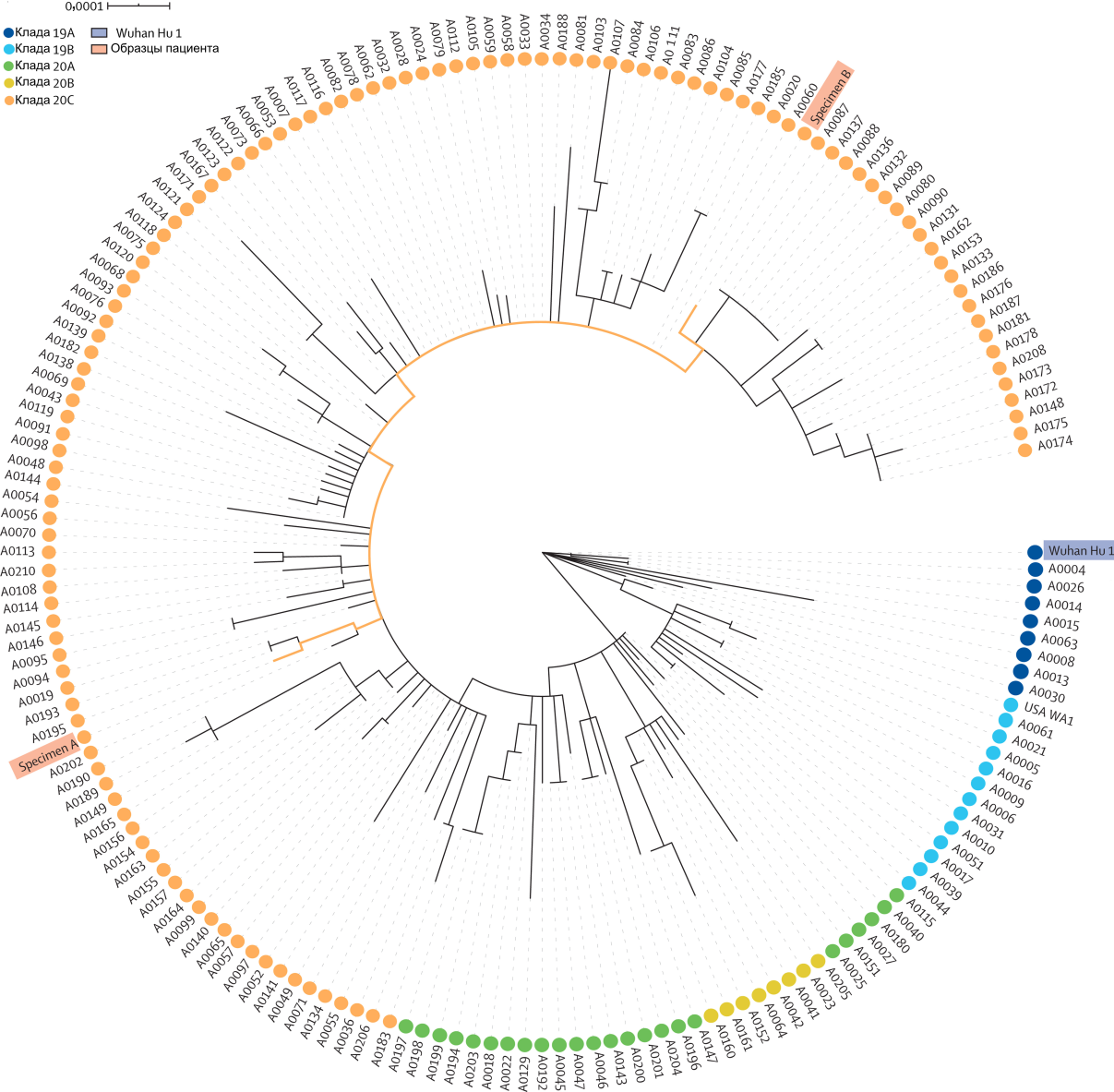
Образцы A и B вошли в число 171 образцов, полученных в штате Невада (США) в промежутке с 5 марта по 5 июня 2020 г. и подвергнутых секвенированию. Филогенетический анализ показывает родство образцов A и B друг с другом и их сравнительное расстояние до других положительных образцов (Рис. 3). Чтобы исключить ошибку при обработке образцов или подписи пробирок во время выделения РНК, мы провели тесты судебно-медицинской идентификации личности для определения принадлежности исходного и промежуточного материалов образцов A и B. Анализ каждого из образцов, а также материалов, оставшихся при выделении и аликвотировании, показал принадлежность образцов A и B одному и тому же человеку (вероятность, что эти образцы принадлежат разным людям, составляет 1:53,48×1024).
Рис. 3. Филогенетическое положение образцов A и B среди образцов из штата Невада, референсных геномов и глобальных клад.
Обсуждение
Мы представляем отчёт о первом пациенте в Северной Америке с сопровождающейся симптомами повторной инфекцией SARS-CoV-2. Наше наблюдение сходно со случаем повторной инфекции в Эквадоре [12] в том, что заболевание при повторной инфекции протекало тяжелее, в то время как в описанных случаях из Бельгии и Нидерландов [11] и из Гонконга [10] тяжесть симптомов не различалась. Механизмы, объясняющие более тяжёлое течение заболевания при реинфекции, можно только предположить. Во-первых, к повторной инфекции и тяжелому течению могла привести очень высокая вирусная нагрузка [14]. Во-вторых, возможно, повторная инфекция была вызвана более вирулентным вариантом вируса или вирусом, более вирулентным для конкретного пациента. В-третьих, причиной мог стать механизм антителозависимого усиления, явление, при котором специфические иммунные клетки с Fc-рецептором становятся инфицированными в результате связывания со специфическими антителами. Этот механизм уже наблюдали в случае бетакоронавируса, вызывающего тяжелый острый респираторный синдром [15]. В упомянутом случае пациент поправился и был выписан.
У пациента с описываемой повторной инфекцией SARS-CoV-2 не было иммунологических заболеваний, которые могли бы облегчить реинфицирование. Пациент не принимал никаких иммуносупрессоров, был ВИЧ-негативным по результатам тестирования на антитела и РНК (данные не показаны) и не имел никаких очевидных отклонений в составе клеток крови. Второй случай заражения (реинфекция) произошел одновременно с заболеванием человека, проживавшего на той же площади (родителя), который также предоставил образец 5 июня 2020 г., и этот образец показал положительный результат при тестировании с помощью амплификации нуклеиновых кислот (транскрипционно-опосредованной амплификации, ТОА). Сейчас проводится секвенирование этого образца, чтобы установить его роль в повторной инфекции. Следует отметить, однако, что положительный образец проживающего совместно с пациентом был получен и протестирован в формате Hologic Aptima, который не соответствует процедурам, принятым в нашей лаборатории, занимающейся секвенированиям. Тем не менее, случай инфекции у совместно проживающего предоставляет возможный источник вторичного заражения и повторного заболевания нашего пациента.
Возможно, что представленный случай представляет собой продолжающуюся инфекцию, включающую деактивацию и реактивацию. Тем не менее, для реализации этой гипотезы скорость мутирования SARS-CoV-2 должна быть намного выше, чем зарегистрированная когда-либо ранее [16, 17, 18, 19]. Экстраполяция замен в образцах A и B соответствует скорости накопления ОНВ и МНВ 83,64 замен в год, что существенно выше наблюдаемой сейчас скорости 23,12 [19]. Ещё важнее, что четыре замены в образце A должны были подвергнуться обратной мутации к предковому генотипу, и вероятность такого события ничтожна. Разумеется, если в упомянутые сроки действительно произошло такое количество нуклеотидных замен, необычность образцов A и B из примера возможной повторной инфекции превратится в пример чрезвычайно высокой скорости эволюции в теле инфицированного. Другим возможным объяснением наблюдаемых различий образцов A и B является коинфекция. В этом случае можно предположить, что пациент был инфицирован вирусами обоих генотипов в момент взятия образца. Эта гипотеза требует предположить, что образец вируса типа B уже присутствовал в апреле 2020 г., но не был обнаружен, и наоборот, количество вирионов типа A уменьшилось к моменту взятия образца в июне 2020 г.. Оба образца, A и B, принадлежат к кладе, наиболее распространённой на севере Невады в период взятия образцов. Наше исследование вирусов из Невады позволило найти образцы, похожие на каждый из обсуждаемых генотипов [20]. Хотя есть свидетельства, что квазивиды SARS-CoV-2 в образцах от инфицированных существуют с низкой и сильно варьирующей частотой [21], т.е. редкие ОНВ (например, с частотой 1%) могут быть обнаружены в разных образцах одного и того же пациента, возможность этой ситуации сама по себе не объясняет смену генотипа между первой и повторной инфекцией.
Наши результаты могут быть важны для понимания роли вакцинации в борьбе с COVID-19. Если описанный случай действительно представляет собой пример реинфекции, первичная экспозиция SARS-CoV-2 может не приводить к уровню иммунитета, который бы давал 100% защиту для каждого человека. Такие сложности вакцинации известны по частым проблемам с эффективным дизайном вакцин от гриппа [22].
Существенным ограничением представленного исследования является отсутствие какой-либо оценки иммунного ответа при первой инфекции SARS-CoV-2. Мы также не смогли полностью оценить эффективность иммунного ответа (например, титры нейтрализующих антител) при втором эпизоде инфекции, когда у пациента определялись титры антител к белку нуклеокапсида SARS-CoV-2. Если описанный случай - пример естественной эволюции вируса in vivo (что крайне маловероятно с учётом необходимости четырёх обратных замен к референсному генотипу), то SARS-CoV-2 имеет достаточную генетическую пластичность и способность к адаптации, чтобы избегать естественного иммунного ответа и восстанавливаться до определяемого уровня у того же пациента. Если описываемый случай объясняется реинфекцией, критически важно, что частота такого события не может быть определена в единственном исследовании: оно может быть редким. Отсутствие полномасштабного геномного секвенирования положительных случаев в США и по всему миру замедляет развитие эпидемиологического надзора, необходимого для обнаружения других таких случаев. Несомненно, ограничения в области скрининга и возможностей тестирования SARS-CoV-2 усугубляют недостаточность надзора, не только для диагностики COVID-19, но и для действенного генетического отслеживания этого возбудителя.
Список литературы
- Ju B, Zhang Q, Ge J et al. Human neutralizing antibodies elicited by SARS-CoV-2 infection. Nature. 2020; 584: 115-119. Crossref
- Callow KA, Parry HF, Sergeant M, Tyrrell DA. The time course of the immune response to experimental coronavirus infection of man. Epidemiol Infect. 1990; 105: 435-446. Crossref
- Chang S-C, Wang J-T, Huang L-M et al. Longitudinal analysis of severe acute respiratory syndrome (SARS) coronavirus-specific antibody in SARS patients. Clin Diagn Lab Immunol. 2005; 12: 1455-1457. Crossref
- Huang AT, Garcia-Carreras B, Hitchings MDT et al. A systematic review of antibody mediated immunity to coronaviruses: antibody kinetics, correlates of protection, and association of antibody responses with severity of disease. medRxiv.2020; (published online April 17.) (preprint) doi.org
- Liu W, Fontanet A, Zhang P-H et al. Two-year prospective study of the humoral immune response of patients with severe acute respiratory syndrome. J Infect Dis. 2006; 193: 792-795. Crossref
- Mo H, Zeng G, Ren X et al. Longitudinal profile of antibodies against SARS-coronavirus in SARS patients and their clinical significance. Respirology. 2006; 11: 49-53. Crossref
- Reed SE. The behaviour of recent isolates of human respiratory coronavirus in vitro and in volunteers: evidence of heterogeneity among 229E-related strains. J Med Virol. 1984; 13: 179-192. Crossref
- Woo PCY, Lau SKP, Wong BHL et al. Longitudinal profile of immunoglobulin G (IgG), IgM, and IgA antibodies against the severe acute respiratory syndrome (SARS) coronavirus nucleocapsid protein in patients with pneumonia due to the SARS coronavirus. Clin Diagn Lab Immunol. 2004; 11: 665-668. Crossref
- Wu L-P, Wang N-C, Chang Y-H et al. Duration of antibody responses after severe acute respiratory syndrome. Emerg Infect Dis. 2007; 13: 1562-1564. Crossref
- To KK-W, Hung IF-N, Ip JD et al. COVID-19 re-infection by a phylogenetically distinct SARS-coronavirus-2 strain confirmed by whole genome sequencing.
Clin Infect Dis. 2020; (published online Aug 25.) doi.org - Van Elslande J, Vermeersch P, Vandervoort K et al. Symptomatic SARS-CoV-2 reinfection by a phylogenetically distinct strain. Clin Infect Dis. 2020; (published online Sept 5.) doi.org
- Prado-Vivar B, Becerra-Wong M, Guadalupe JJ et al. COVID-19 re-infection by a phylogenetically distinct SARS-CoV-2 variant, first confirmed event in South America.
SSRN. 2020; (published online Sept 8.) (preprint) doi.org
Crossref - Abnizova I, te Boekhorst R, Orlov YL. Computational errors and biases in short read next generation sequencing. J Proteomics Bioinform. 2017; (published online Jan 26.)
doi.org - Guallar MP, Meiriño R, Donat-Vargas C, Corral O, Jouvé N, Soriano V. Inoculum at the time of SARS-CoV-2 exposure and risk of disease severity. Int J Infect Dis. 2020; 97: 290-292. Full Text
- Yip MS, Leung NH, Cheung CY et al. Antibody-dependent infection of human macrophages by severe acute respiratory syndrome coronavirus. Virol J. 2014; 11: 82. Crossref
- Hill V, Rambaut A. Phylogenetic analysis of SARS-CoV-2 genomes. virological.org
Date: March 6, 2020. Date accessed: September 23, 2020 - Mercatelli D, Giorgi FM. Geographic and genomic distribution of SARS-CoV-2 mutations. Front Microbiol. 2020; 111800. Crossref
- Pachetti M, Marini B, Benedetti F et al. Emerging SARS-CoV-2 mutation hot spots include a novel RNA-dependent-RNA polymerase variant. J Transl Med. 2020; 18: 179. Crossref
- Hadfield J, Megill C, Bell SM et al. Genomic epidemiology of novel coronavirus: global subsampling. nextstrain.org Date: Sept 16, 2020 Date accessed: September 23, 2020
- Hartley P, Tillett RL, Xu Y et al. Genomic surveillance revealed prevalence of unique SARS-CoV-2 variants bearing mutation in the RdRp gene among Nevada patients.
medRxiv.2020; (published online Sept 11.) (preprint) doi.org - Jary A, Leducq V, Malet I et al. Evolution of viral quasispecies during SARS-CoV-2 infection. Clin Microbiol Infect. 2020; (published online July 24.) Full Text
- Osterholm MT, Kelley NS, Sommer A, Belongia EA. Efficacy and effectiveness of influenza vaccines: a systematic review and meta-analysis.
Lancet Infect Dis. 2012; 12: 36-44. Full Text